PCR (Polymerase Chain Reaction)
Type de laboratoire |
Type de technique |
Objectif |
Bioagresseur(s) concerné(s) |
Temps de mise en oeuvre |
Délai de réponse du laboratoire |
|
Laboratoire de détection Laboratoire spécialisé |
Moléculaire |
Détection |
Bactéries, Champignons, Virus, Phytoplasmes, Nématodes |
1 - 2 jours |
8 - 15 jours ouvrés |
Principe :
Cette technique a été inventée en 1983 par K. Mullis et brevetée en 1985. C’est une technique qui permet de détecter et de quantifier un bioagresseur par son matériel génétique. La PCR repose sur l’amplification spécifique d’une séquence d’acide nucléique in vitro. Chaque cycle de PCR s’effectue en 3 étapes : dénaturation thermique de l’ADN à 95°C, hybridation des amorces à 50-65°C, élongation à 72°C.
- PCR classique
Les différentes étapes sont réalisées à plusieurs températures dans un appareil appelé thermocycleur. Le mélange réactionnel est placé dans des microtubes et est soumis, plusieurs dizaines de fois, à des cycles successifs. L’appareil permet la programmation de chaque étape des cycles. La réaction PCR est extrêmement rapide, elle ne dure que quelques heures (2 à 3 heures pour une PCR de 30 cycles). Elle est réalisée dans un mélange réactionnel constitué de : Taq polymérase, amorces, les 4 dNTP en excès (A, T, C, G) et l’extrait d’ADN cible.
Les étapes sont :
1. Cycle de PCR. Il s'effectue en 3 étapes :
- La dénaturation thermique de l'ADN à 95°C. Les 2 brins de l'ADN se séparent par rupture des liaisons hydrogènes. Nous obtenons alors des matrices simple brin.
- Hybridation des amorces à 50°C-65°C. Les deux amorces contenues en large excès s’hybrident lorsqu’elles rencontrent les séquences complémentaires. La température permet aux liaisons hydrogènes de se reformer. Les amorces s’hybrident plus facilement que les longs brins d’ADN ainsi, plus la température d’hybridation est élevée, plus l’hybridation est sélective et spécifique.
- Elongation à 72°C. La Taq polymérase (ADN polymérase) catalyse la réplication à partir des ADN monocaténaires amorcés donc de façon sélective (sélectivité qui découle du choix des amorces).
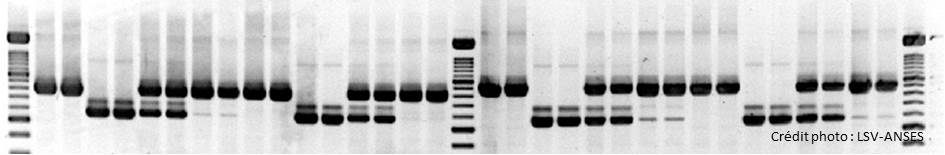
2. Electrophorèse sur gel d’agarose ou d’acrylamide. Cette étape repose sur l’attraction des acides nucléiques chargés négativement sous l’effet d’un champ électrique. Les acides nucléiques migrent à travers le gel avec des vitesses différentes selon la masse de la molécule.
3. Révélation. L’ADN est révélé par une coloration au Bromure d’éthidium ou plus récemment avec du SYBR®Green visible sous une lampe UV.
- PCR en temps réel
La PCR en temps réel est basée sur une PCR classique à laquelle est ajoutée une mesure de l’amplification tout au long de la réaction d’où le terme « en temps réel ». Elle repose sur la détection et la quantification par un signal fluorescent.
Les étapes sont :
1. Cycle PCR :
- Dénaturation thermique de l'ADN à 95°C
- Hybridation des amorces à 50°C-65°C
- Elongation à 72°C
2. Révélation. La révélation est réalisée grâce à une courbe de fluorescence. La valeur de la fluorescence est corrélée à la quantité de produit amplifié.
3. Résultats et quantification
- RT-PCR
La RT-PCR permet de faire une PCR à partir d’ARN qui est rétrotranscrit par une transcriptase inverse en ADN complémentaire (ADNc). Ce dernier est utilisé pour réaliser une PCR.
Les étapes sont :
1. Transcription inverse : ARN en ADNc
2. Cycle PCR
- Dénaturation thermique de l'ADN à 95°C
- Hybridation des amorces à 50°C-65°C.
- Elongation à 72°C
3. Electrophorèse
4. Révélation UV
- Séquençage des produits PCR
Si la PCR n'est pas assez spécifique pour identifier le bioagresseur, il est possible de réaliser un séquençage des produits obtenus afin d'affiner le diagnostic.
Avantages |
Inconvénients |
|
|